Rを使った、データの読み込み、フィッティング、データの書き出し
Rでの解析方法についての覚書。
まずは、Rを使ったデータの読み込み方法。
(1) テキストファイルからのデータの読み込み
> data = read.table("C:\\------.txt"(ファイルの指定), header = F)ここでの"data"は読み込んだデータの入れ物。
header = F
これは、第1行に変数名がないことを指定している。(もし、変数名がある場合は”T”にする)
このコードでは、"C:\\------.txt"のように開くファイルをタイプする必要がある。(打ち間違えのリスクもあり面倒)
そのため、
> data = read.table(file.choose(), header = F)
のようにfile.choose()を使用すると、「ファイルを開く」ダイアログが表示されファイルを選択できるようになり便利。
> data = read.table(file.choose(), header = F)
(2) Excelファイルからのデータの読み込み
(a) Excelファイルを指定して、データを読み込む
まず下準備として、Rの“パッケージ”から“パッケージのインストール”をクリック。Packagesの中から“xlsx”を選択。
> library (xlsx) > data2 = read.xlsx(file.choose(), 1, stringsAsFactors = F, header = F)
Excelファイルを開くには、
> library (xlsx)
で、“xlsx”を呼び込む必要がある。(Rの起動時毎に)
途中にある“1”はシート番号、第1行に変数名がない場合は“stringsAsFactors = F, header = F”にしておく。
(b) Excelファイル上の特定のデータを読み込む
まず下準備として、Excelファイル上の解析したいデータをコピーしておく。
> data3 = read.table("clipboard", header = F)このようにして、Excelファイル上の特定のデータを読み込む場合は、“library (xlsx)”の必要はない。
読み込んだデータは、
> data
で、確認できる。
ここでは、次のようなデータを読み込んだ。
V1 V2 1 12 0 2 10 1 3 8 2 4 11 3 5 6 4 6 7 5 7 2 6 8 3 7 9 3 8
次に、読み込んだデータをグラフにする。
> x = data[[1]] > y = data[[2]] > attach (data) > plot (x, y, pch = 16)
このコードで、下記のようなグラフが書ける。
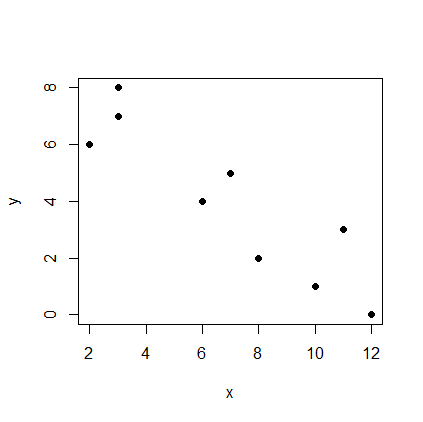
> x = data[[1]] > y = data[[2]]
では、取り込んだデータの1列目を“x”、2列目を“y”としている。
> plot (x, y, pch = 16)
この部分で、x-yのグラフを描かせ、表示を黒丸(pch = 16)にしている。
読み込んだデータの線形回帰。
> fm <- lm (y ~ x) > abline(fm)
このコードにより下記のように回帰直線を求めて引くことが出来る。
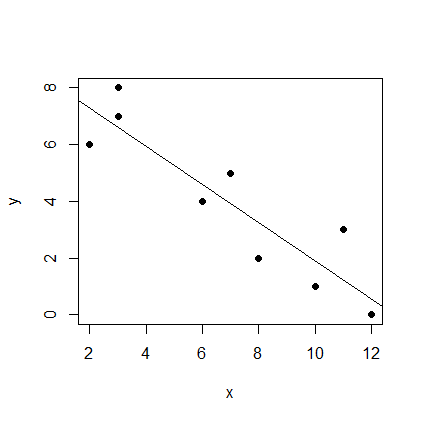
lm (y ~ x)
この部分で、どの変数が応答変数で(ここではy)、どの変数が説明変数(ここではx)なのかを、Rに教える。
線形回帰の場合は“lm”を用いて、非線形回帰の場合は“nls”を使用する。
線形回帰の結果は
> summary (fm))
で呼び出すことができ、ここでは下記の様になった。
Call:
lm(formula = y ~ x)
Residuals:
Min 1Q Median 3Q Max
-1.2775 -0.9143 -0.5735 1.0745 1.7561
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 8.6184 0.9296 9.271 3.52e-05 ***
x -0.6704 0.1205 -5.565 0.000846 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 1.257 on 7 degrees of freedom
Multiple R-squared: 0.8157, Adjusted R-squared: 0.7893
F-statistic: 30.97 on 1 and 7 DF, p-value: 0.0008461最後に、解析結果をエクセル等に書き出す。
> names(fm)
で、解析結果の中身を調べることが出来、結果が下記。
[1] "coefficients" "residuals" "effects" "rank" [5] "fitted.values" "assign" "qr" "df.residual" [9] "xlevels" "call" "terms" "model"
この中で、[1]"coefficients"が回帰直線の傾きと切片を示し、
> fm$coefficients
上記のコードで、下の様に、その値のみを呼び出すことが出来る。
(Intercept) x 8.6183673 -0.6704082
解析結果は、
> write.xlsx(fm$coefficients, file.choose(), col.names = T, row.names = T)
とすることで、excelファイルとして保存できる。ここでは、
file.choose()
を使用しているため、任意の名前で保存できる。(但し、拡張子(.xlsx)を付け忘れないように)
col.names = T, row.names = T
この部分は、行と列の名前を出力するかどうかを指定する箇所で、出力しない場合は“F”にする。
最後に、コードの全体を書く。
> library (xlsx) > data = read.xlsx(file.choose(), 1, stringsAsFactors = F, header = F) > attach (data) > x = data[[1]] > y = data [[2]] > plot (x, y, pch = 16) > fm <- lm(y ~ x) > abline(fm) > write.xlsx(fm$coefficients, file.choose(), col.names = T, row.names = T)
Javaで書くよりもかなりスッキリしている。